2022年2月12日,《Bioinformatics》在线发表了题为《Multimodal reasoning based on knowledge graph embedding for specific diseases》的论文,该文提出了一个完整的多模态专病知识图谱的构建及推理流程。刘雷课题组博士生朱超宇和夏晓琼为本文第一和第三作者,大连理工大学杨志豪教授和博士生李楠为第二和第四作者,我院刘雷研究员和钟凡副教授为共同通讯作者。

知识图谱(Knowledge Graph,KG)是知识的一种存储方式,它通过海量三元组(头实体,关系,尾实体)表示现实世界中的事实。KG在生物医学领域广泛用于预测药物靶点相互作用和药物不良反应,以及临床决策支持系统的构建。
现有的生物医学KG主要关注于某一实体,而鲜有工作聚焦于某一专病。本文在这一工作中考虑了11种专病,包括5种癌症以及6种非癌疾病。以关键词从PubMed收集相关摘要后,杨志豪团队通过命名实体识别和关系提取获得原始三元组,经过实体和关系的细粒度匹配,得到SDKG-11,它可以表示这些专病现有的知识合集。
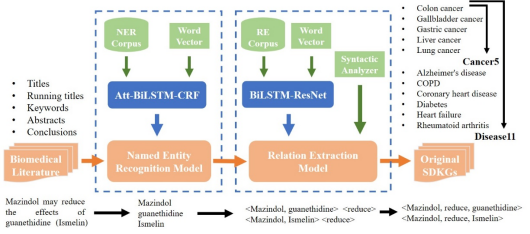
但是新的生物医学知识每天都在被不停提出,已构建完成的KG几乎都是不完整的。因此,从现有KG推理可靠的新知识是一个普遍的想法。知识图谱嵌入(Knowledge Graph Embedding,KGE)将实体和关系映射到一个连续向量空间,用简单的数字计算取代复杂的推理过程,已经成为KG推理的一个范式。刘雷团队将KGE结构、类别和描述三种模态的嵌入通过反式超平面投影的方式进行结合,定义了多模态评分函数。

通过定义的评分函数,他们得到了排名靠前的药物-基因,基因-疾病,疾病-药物实体对。然后经过文献证据的搜索,他们验证了新推理知识的可靠性。通过将嵌入结果作为生物分子相互作用分类任务的预训练初始化参数,他们验证了嵌入结果的普适性。本文希望针对专病知识图谱进行多模态推理,生成对科研探索和临床决策有指导价值的新知识集合。